在 Matlab 中,我想制作一个氨基酸序列图谱的seqlogo 图。但不是通过熵来缩放绘图列的高度,而是希望所有列的高度相同。
我正在根据这个问题的答案修改代码,但我想知道是否有 seqlogo 的参数或我错过的其他一些函数会使列高统一。
或者,我是否可以将统计转换应用于序列配置文件以破解所需的输出?(列高度一致,每个字母的高度与其在 seqprofile 中的概率成线性比例)
在 Matlab 中,我想制作一个氨基酸序列图谱的seqlogo 图。但不是通过熵来缩放绘图列的高度,而是希望所有列的高度相同。
我正在根据这个问题的答案修改代码,但我想知道是否有 seqlogo 的参数或我错过的其他一些函数会使列高统一。
或者,我是否可以将统计转换应用于序列配置文件以破解所需的输出?(列高度一致,每个字母的高度与其在 seqprofile 中的概率成线性比例)
解决此问题的最简单方法可能是直接修改Bioinformatics Toolbox 函数 SEQLOGO的代码(如果可能)。在 R2010b 中,您可以执行以下操作:
edit seqlogo
该函数的代码将显示在编辑器中。接下来,找到以下行(第 267-284 行)并将它们注释掉或完全删除它们:
S_before = log2(nSymbols);
freqM(freqM == 0) = 1; % log2(1) = 0
% The uncertainty after the input at each position
S_after = -sum(log2(freqM).*freqM, 1);
if corrError
% The number of sequences correction factor
e_corr = (nSymbols -1)/(2* log(2) * numSeq);
R = S_before - (S_after + e_corr);
else
R = S_before - S_after;
end
nPos = (endPos - startPos) + 1;
for i =1:nPos
wtM(:, i) = wtM(:, i) * R(i);
end
然后将这一行放在他们的位置:
wtM = bsxfun(@times,wtM,log2(nSymbols)./sum(wtM));
您可能希望以新名称保存文件,例如seqlogo_norm.m,因此您仍然可以使用原始未修改的SEQLOGO函数。现在,您可以创建所有列标准化为相同高度的序列剖面图。例如:
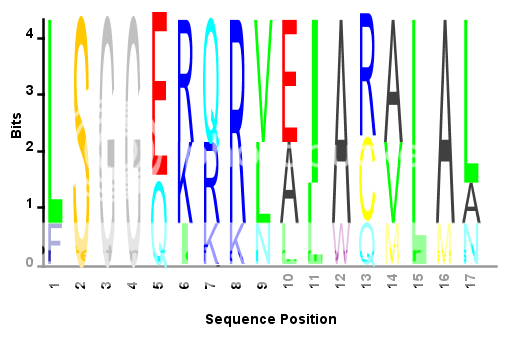
S = {'LSGGQRQRVAIARALAL',... %# Sample amino acid sequence
'LSGGEKQRVAIARALMN',...
'LSGGQIQRVLLARALAA',...
'LSGGERRRLEIACVLAL',...
'FSGGEKKKNELWQMLAL',...
'LSGGERRRLEIACVLAL'};
seqlogo_norm(S,'alphabet','aa'); %# Use the modified SEQLOGO function
旧答案:
我不确定如何转换序列配置文件信息以从Bioinformatics Toolbox 函数 SEQLOGO获得所需的输出,但我可以向您展示如何修改我为回答您链接到的相关问题seqlogo_new.m而编写的替代方案。如果您更改从此初始化的行:bitValues
bitValues = W{2};
对此:
bitValues = bsxfun(@rdivide,W{2},sum(W{2}));
然后,您应该将每列缩放到 1 的高度。例如:
S = {'ATTATAGCAAACTA',... %# Sample sequence
'AACATGCCAAAGTA',...
'ATCATGCAAAAGGA'};
seqlogo_new(S); %# After applying the above modification

目前,我的解决方法是生成一堆与序列配置文件匹配的假序列,然后将这些序列提供给http://weblogo.berkeley.edu/logo.cgi。这是制作假序列的代码:
function flatFakeSeqsFromPwm(pwm, letterOrder, nSeqsToGen, outFilename)
%translates a pwm into a bunch of fake seqs with the same probabilities
%for use with http://weblogo.berkeley.edu/
%pwm should be a 4xn or a 20xn position weight matrix. Each col must sum to 1
%letterOrder = e.g. 'ARNDCQEGHILKMFPSTWYV' for my data
%nSeqsToGen should be >= the # of pixels tall you plan to make your chart
[height windowWidth] = size(pwm);
assert(height == length(letterOrder));
assert(isequal(abs(1-sum(pwm)) < 1.0e-10, ones(1, windowWidth))); %assert all cols of pwm sum to 1.0
fd = fopen(outFilename, 'w');
for i = 0:nSeqsToGen-1
for seqPos = 1:windowWidth
acc = 0; %accumulator
idx = 0;
while i/nSeqsToGen >= acc
idx = idx + 1;
acc = acc + pwm(idx, seqPos);
end
fprintf(fd, '%s', letterOrder(idx));
end
fprintf(fd, '\n');
end
fclose(fd);
end